Análisis de la diversidad genética en genotipos de alfalfa (Medicago sativa L.) sin dormición, mediante el uso de marcadores SSR y caracteres agronómicos
Palabras clave:
autotetraploide, marcadores microsatélites, análisis de clusters, análisis de componentes principale, análisis de procrustes generalizado, mejoramiento vegetalResumen
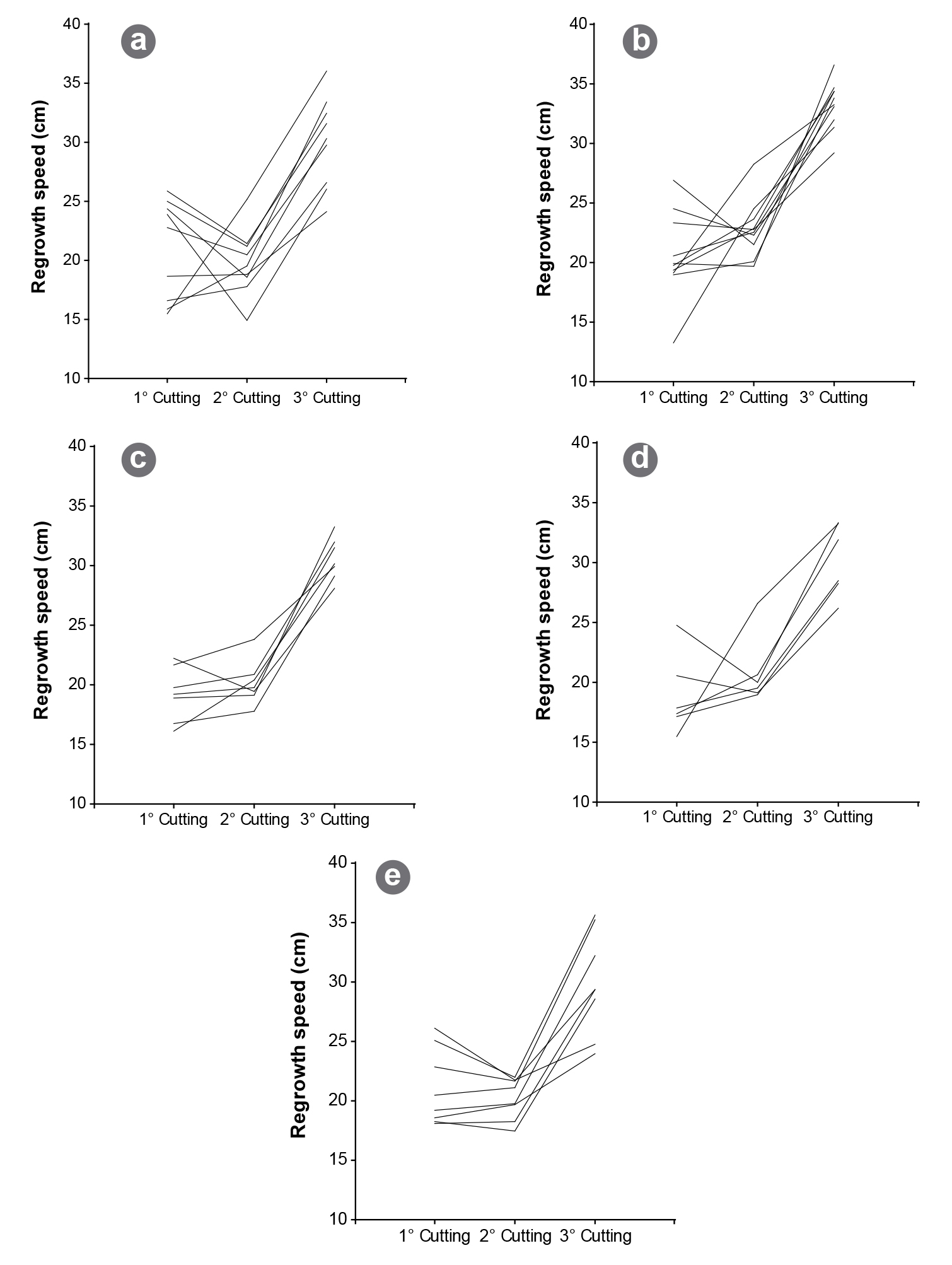
El objetivo de este trabajo consistió en determinar la diversidad genética en 40 individuos de alfalfa (Medicago sativa L.) de cinco cultivares de grado de reposo 8. Se evaluaron a campo la producción de forraje, la velocidad de rebrote y el comportamiento frente a tallo negro de primavera, mancha ocular y roya. El análisis de la varianza se realizó mediante un modelo lineal mixto para determinar la variación agronómica entre los individuos. El análisis de componentes principales se realizó a partir de los datos agronómicos estandarizados. El dendrograma agronómico se construyó a partir del índice de Gower y el agrupamiento UPGMA. Para la caracterización molecular se analizaron seis marcadores SSR, con los cuales se identificaron un total de 84 alelos. Las distancias genéticas se calcularon con el índice de Nei estándar. La diversidad genética promedio fue de 0,843, indicando una alta variabilidad entre los individuos evaluados. Por último, el análisis de procrustes generalizado detectó un 65,4% de consenso entre el ordenamiento agronómico y el molecular. Este porcentaje probablemente se relacione con el bajo número de individuos y marcadores SSR analizados; sin embargo, este estudio provee una línea de base para estudios futuros de diversidad que permitirán identificar individuos o cultivares genéticamente distantes.
Descargas
Publicado
Número
Sección
Licencia

Esta obra está bajo una licencia internacional Creative Commons Reconocimiento-NoComercial-CompartirIgual 3.0.
Aquellos autores/as que tengan publicaciones con esta revista, aceptan las Políticas Editoriales.



